D3のSさんが環境DNA分析の様子を紹介してくれました!
ーーー
今年の夏に北海道周辺海域とグリーンランドでサンプリングした環境DNAの分析を北海道大学大学院水産科学院で実施してきました.環境DNA(environmental DNA, eDNA)とは,湖沼や河川,海洋などの水中に存在する生物の由来のDNA断片のことです(Fig 1).環境DNAを用いて,魚類などのマクロ生物の分布や生物量推定に利用されています.
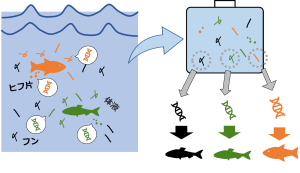
Fig 1. 環境DNAはその環境に生息する生物の排泄物や粘液,はがれた皮膚片などに由来するDNA断片のことです.環境DNAの調査では水をくみ取ることでこれらのDNAを採取し,そのDNAから生物種を推定します.
今回,環境DNA分析で扱うサンプルは今年の夏に北海道東部知床半島周辺海域やグリーンランドのフィヨルドで採水したものです.みなさんもご存知の通り,これらの地域には数多くの海棲哺乳類が生息しています.では,彼らがなぜそこに集まるのでしょうか,その要因を探るために,今回は彼らの餌生物である魚類に着目し,海棲哺乳類の集まる場所の魚類相にはどのような特徴があるのかを環境DNAで明らかにすることを目的としています.
まずはeDNAを含む海水の採水です.採水にはニスキン採水器を用いました.北海道周辺海域では,北海道大学練習船おしょろ丸に乗船し,知床半島周辺海域で海面から海底直上までの間の複数深度で採水しました.おしょろ丸にはFig 2aのように複数の採水器が装備されており,スムーズに採水ができます.一方で,グリーンランドのフィヨルドには数多くの氷山が存在するため,大型調査船の侵入が難しいです.そのため,現地住民のボートにウィンチを取り付け,目的の地点まで移動して採水していきます(Fig 2b).そして,これらのニスキンン採水器で採水した海水は採水パックに詰めて濾過をする場所まで運びます.


次は採水した海水の濾過です(Fig 3).濾過にはステリベクスを使用しました.魚のDNAのコンタミネーションに気をつけるため,積極的にハイターで作業場所を拭き,作業を進めました.そして,環境DNAがフィルターされたステリベクスはRNAlater添加後,次の分析まで冷凍保存しました.

Fig 3. おしょろ丸で海水を濾過している様子.採水袋の口に直接ステリベクスが取り付けられ,その先にチューブを取り付けてアスピレーターで濾過をした.
ここからの作業は,北海道大学大学院水産科学院で実施しました(Fig 4).冷凍しておいたステリベクスは解凍し,DNA抽出キッドを用いて抽出しました(Fig 5).DNA抽出産物は次の作業までまた冷凍保存をします.

Fig 4. 北海道大学大学院水産科学院の校門.12月の函館は雪がたくさん積もっていました.
続いて, PCRを2回実施します.1st PCRではサンプルにMiFishプライマーを加えて,魚類全般のDNAを増幅させます.次に2nd PCRではシーケンス時にサンプルを識別するためのインデックス配列を付加します.そして,電気泳動をして各サンプルのバンドの明るさからサンプル濃度を判断します.細かい作業が続きます...
そして,判断された濃度を元にPCR産物を混合し,シーケンスライブラリの作成をします.ここでは,Ampure XPを用いたビーズ精製を行いました.個人的に,ビーズの入った茶色の混合液がだんだんと磁気によって透明になっていく様子は見ていて楽しく感じました(Fig 5).

Fig 5. ビーズ精製の様子.(a)は最初の様子,(b)は磁気によって寄せられ,透明になった様子.
そして,E-Gelによって目的のバンドのDNAを回収し(Fig 6),Qubitによる濃度測定を行って,バイオアナライザで濃度測定したサンプルの精製度をチェックしました.個人的に,ここのステップでは,ちゃんと目的のDNAを上手く回収できるかドキドキしながら進めました...

Fig 6. E-Gelで目的のバンドのDNAを抽出.
そして,いよいよシーケンスです.まず,使用するサンプルの濃度を薄めていきます.40 pMとか,普段あまり見かけないような単位に薄める作業だったので,ここも若干ドキドキしながら作業しました.
そして,調整したサンプルをiSeq(Fig 7)に入れて,配列を読んでいきます.シーケンスは約18時間かかるため,その間もちゃんとデータが得られるか,ドキドキしていました.
…環境DNAの分析中,ずっとドキドキしていますね(笑)

Fig 7. 使用したiSeq.結果が楽しみです!
最終的に,今回分析したサンプルすべてを調べることができました.現在,得られた配列の解析を進めております.みなさんに結果を見せるのがとても楽しみです.
最後に,今回,環境DNAの分析をさせていただいた北海道大学大学院水産科学院の笠井先生と川上達也 博士をはじめ,同研究室の皆様には大変お世話になりました.この場をお借りして感謝申し上げます.ありがとうございました!!
海洋環境科学講座笠井研究室 HP: https://lab-kasai-jp.jimdosite.com/